caretで独自アルゴリズムの実行
caretパッケージは,機械学習のモデル構築・評価を統一したフレームワークで実行するための機能を提供している.caretのバージョン6.0.29では150個のアルゴリズムが利用できる.
> library(caret) > packageVersion("caret") [1] ‘6.0.29’ > head(modelLookup()) model parameter label forReg forClass probModel 1 ada iter #Trees FALSE TRUE TRUE 2 ada maxdepth Max Tree Depth FALSE TRUE TRUE 3 ada nu Learning Rate FALSE TRUE TRUE 4 avNNet size #Hidden Units TRUE TRUE TRUE 5 avNNet decay Weight Decay TRUE TRUE TRUE 6 avNNet bag Bagging TRUE TRUE TRUE > length(unique(modelLookup()[, "model"])) [1] 150
ユーザは,caretで提供されているアルゴリズム以外のものも追加できる.そのためにはまず, caretで実装されているモデルがどのような情報を含んでいるかについて理解する必要がある.
> # サポートベクタマシン (RBF カーネル ) の情報を取得 > svmRadial.ModelInfo <- getModelInfo(model = "svmRadial", regex = FALSE)[[1]] > names(svmRadial.ModelInfo) [1] "label" "library" "type" "parameters" "grid" [6] "loop" "fit" "predict" "prob" "predictors" [11] "tags" "levels" "sort"
以上の結果を見ると,13個の情報を保持していることが分かる.これらを含めて,14個の情報を保持できる.それぞれの意味を下表に示す.
| パラメータ | 内容 | データ型 |
|---|---|---|
| library | 【必須】予測モデルのフィッティングや予測に用いるパッケージの名称 | character |
| type | 【必須】予測の種別. "Classification":クラス分類, "Regression":回帰, もしくは両方を指定 | character |
| parameters | 【必須】ハイパーパラメータの名前,型,名称 | data.frame |
| grid | 【必須】ハイパーパラメータの探索範囲を生成する関数 | function |
| fit | 【必須】モデルをフィッティングする関数 | function |
| predict | 【必須】予測を行う関数 | function |
| prob | 【必須】クラス確率を算出する関数 | function |
| sort | 【必須】ハイパーパラメータを最も複雑なものからソートする関数 | function |
| loop | 【任意】予測モデルが複数のサブモデルの予測を返す場合に指定する関数 | function |
| levels | 【任意】クラス分類モデルに対して,予測値のカテゴリ水準を返す関数 | function |
| tags | 【任意】予測モデルに関連するタグ(例:"Tree-Based Model","Embedded Feature Selection") | character |
| label | 【任意】予測モデルのラベル(例:"Linear Discriminant Analysis") | character |
| predictors | 【任意】説明変数の名前のベクトル | character |
| varImp | 【任意】説明変数の重要度を算出する関数 | function |
caretは,ラプラスカーネルのサポートベクタマシンは提供していない.caretのホームページの説明を参考に,このアルゴリズムを追加してみよう.
> # ラプラスカーネルのサポートベクタマシンを追加するための設定 > ## 1. 新規リストの作成 > svmLP <- list(type = "Classification", library = "kernlab", loop = NULL) > ## 2. ハイパーパラメータの指定 > prm <- data.frame(parameter = c("C", "sigma"), class = rep("numeric", 2), + label = c("Cost", "Sigma")) > prm parameter class label 1 C numeric Cost 2 sigma numeric Sigma > svmLP$parameters <- prm > ## 3. ハイパーパラメータのデフォルトの探索範囲の指定 > svmGrid <- function(x, y, len = NULL) { + library(kernlab) + sigmas <- sigest(as.matrix(x), na.action = na.omit, scaled = TRUE) + expand.grid(sigma = mean(sigmas[-2]), C = 2^((1:len) - 3)) + } > svmLP$grid <- svmGrid > ## 4. フィッティング関数の指定 > svmFit <- function(x, y, wts, param, lev, last, weights, classProbs, ...) { + ksvm(x = as.matrix(x), y = y, kernel = rbfdot, + kpar = list(sigma = param$sigma), C = param$C, + prob.model = classProbs, ...) + } > svmLP$fit <- svmFit > ## 5. 予測を実行する関数の指定 > svmPred <- function(modelFit, newdata, preProc = NULL, submodels = NULL) { + predict(modelFit, newdata) + } > svmLP$predict <- svmPred > ## 6. クラス確率を推定する関数の指定 > svmProb <- function(modelFit, newdata, preProc = NULL, submodels = NULL) { + predict(modelFit, newdata, type = "probabilities") + } > svmLP$prob <- svmProb > ## 7. ハイパーパラメータをソートする関数の指定 > svmSort <- function(x) x[order(x$C), ] > svmLP$sort <- svmSort > ## 8. クラスの水準の指定 > svmLP$levels <- function(x) lev(x) > # 設定の確認 > svmLP $type [1] "Classification" $library [1] "kernlab" $loop NULL $parameters parameter class label 1 C numeric Cost 2 sigma numeric Sigma $grid function (x, y, len = NULL) { library(kernlab) sigmas <- sigest(as.matrix(x), na.action = na.omit, scaled = TRUE) expand.grid(sigma = mean(sigmas[-2]), C = 2^((1:len) - 3)) } $fit function (x, y, wts, param, lev, last, weights, classProbs, ...) { ksvm(x = as.matrix(x), y = y, kernel = rbfdot, kpar = list(sigma = param$sigma), C = param$C, prob.model = classProbs, ...) } $predict function (modelFit, newdata, preProc = NULL, submodels = NULL) { predict(modelFit, newdata) } $prob function (modelFit, newdata, preProc = NULL, submodels = NULL) { predict(modelFit, newdata, type = "probabilities") } $sort function (x) x[order(x$C), ] $levels function (x) lev(x)
C50パッケージは,顧客の離反を予測するためのデータセットchurnTrain, churnTestを提供している.ここでは,訓練用のchurnTrainデータセットを用いて,上記で定義したsvmLPを用いて学習してみよう.
library(C50) > head(churnTrain, 3) state account_length area_code international_plan voice_mail_plan 1 KS 128 area_code_415 no yes 2 OH 107 area_code_415 no yes 3 NJ 137 area_code_415 no no number_vmail_messages total_day_minutes total_day_calls 1 25 265.1 110 2 26 161.6 123 3 0 243.4 114 total_day_charge total_eve_minutes total_eve_calls total_eve_charge 1 45.07 197.4 99 16.78 2 27.47 195.5 103 16.62 3 41.38 121.2 110 10.30 total_night_minutes total_night_calls total_night_charge 1 244.7 91 11.01 2 254.4 103 11.45 3 162.6 104 7.32 total_intl_minutes total_intl_calls total_intl_charge 1 10.0 3 2.70 2 13.7 3 3.70 3 12.2 5 3.29 number_customer_service_calls churn 1 1 no 2 1 no 3 0 no > # 10-fold クロスバリデーションの実行 > fit.lpSVM <- train(churn ~., data=churnTrain, method=svmLP, + preProc=c("center", "scale"), + trContorl=trainControl(number=10)) > fit.lpSVM 3333 samples 19 predictors 2 classes: 'yes', 'no' Pre-processing: centered, scaled Resampling: Bootstrapped (25 reps) Summary of sample sizes: 3333, 3333, 3333, 3333, 3333, 3333, ... Resampling results across tuning parameters: C Accuracy Kappa Accuracy SD Kappa SD 0.25 0.853 0 0.00712 0 0.5 0.854 0.0198 0.00747 0.0171 1 0.867 0.192 0.00807 0.0471 Tuning parameter 'sigma' was held constant at a value of 0.00747278 Accuracy was used to select the optimal model using the largest value. The final values used for the model were C = 1 and sigma = 0.00747.
高次元データの外れ値検出
高次元データの外れ値検出についてのメモ.
高次元データと次元の呪い
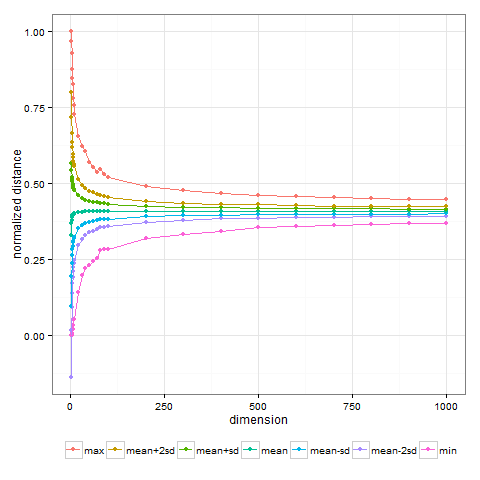
次元が大きくなるほど,点の間の距離は均一になっていく.
例として,2000個の点の各座標を一様乱数で発生させて,次元を変えながら点の間の距離の平均値,最大値,最小値,平均値±1σ,平均値±2σをみてみよう.
library(ggplot2) set.seed(123) # 次元のリスト dims <- c(1:9, 10*(1:9), 100*(1:10)) # 算出する統計量 stats <- c("min", "mean-sd", "mean", "mean+sd", "max") # 発生させる点の個数 N <- 2000 # 各次元に対して算出した統計量を格納する行列 ans <- matrix(NA, length(dims), length(stats), dimnames=list(dims, stats)) # 各次元に対して発生させた点間の距離の統計量を算出する for (d in dims) { message("d=", d) # 点を発生させる(N * d の行列) x <- replicate(d, runif(N)) # 点間の距離を算出する x.dist <- as.matrix(dist(x)) xd <- x.dist[upper.tri(x.dist)] # 点間の距離の統計量を算出する xd.min <- min(xd, na.rm=T) xd.mean <- mean(xd, na.rm=T) xd.max <- max(xd, na.rm=T) xd.sd <- sd(xd, na.rm=T) # 統計量を格納する(次元間を比較できるようにsqrt(dim)で割る) ans[as.character(d), ] <- c(xd.min, xd.mean-xd.sd, xd.mean, xd.mean+xd.sd, xd.max)/sqrt(d) } # 次元と統計量の関係をプロットする ans.m <- melt(ans) ans.m$X2 <- factor(ans.m$X2, levels=rev(stats)) p <- ggplot(data=ans.m, aes(x=Var1, y=value, group=Var2, colour=Var2)) + geom_point() + geom_line() + theme_bw() %+replace% theme(legend.position="bottom", legend.direction="horizontal", legend.title=element_blank()) + xlab("dimension") + ylab("normalized distance") print(p)
確かに次元が高くなるほど点の間の距離は近づいていることが分かる.
この現象は次元の呪い(curse of dimension)に起因しており,高次元において近傍や点の乖離といった概念が意味をなさなくなってしまうことを物語っている.低次元のデータに対しては有効であったデータ間の距離や密度などに基づく外れ値検出が,高次元においては有効ではなくなってしまうのである.
高次元データの外れ値検出のアルゴリズム
このような問題点を解決するために,非常に多くの高次元データの外れ値検出アルゴリズムが提案されている.ここでは,Aggrarwalの"Outlier Analysis",および,Zimek,Schubert,Kriegelによるtutorialを参考に,代表的(と思われる)アルゴリズムをいくつか選び,そのアイディアについて整理する*1.
| 方針 | アルゴリズム | アイディア |
|---|---|---|
| 角度に基づく方法 | ABOD (H.-P.Kriegel, M.Schubert, and A.Zimek, 2008) |
外れ値は,自身と他の2点のデータのなす角度の分散が小さい*2. |
| グリッドによる方法 | Aggarwal and Yu (Aggarwal and Yu, 2001) |
空間をグリッドで分割してデータの個数が少ないグリッドに属する点を外れ値とする. |
| 距離に基づく方法(部分空間への射影) | SOD (H.-P.Kriegel, P.Kroger, E.Schubert, and A.Zimek, 2009) |
各点に対して,k-近傍点の分散が最も小さくなる超平面との距離を外れ値スコアとする. |
| ランダムな部分空間の選択 | Feature Bagging (C.Sutton, M.Sindelar, and A.McCallum, 2005) |
部分空間をランダムに選択する試行を複数回繰り返し,各試行における外れ値スコアの平均値を算出する. |
| コントラストが高い部分空間の選択 | HiCS (F.Keller, E.Muller, and K.Bohm, 2012) |
ある座標の周辺確率密度関数と,他の座標の条件付き確率密度関数が大きく異なる(コントラストが高い)点を外れ値として検出する.このような部分空間を探索し,外れ値スコアを算出する.部分空間の探索と外れ値スコアの算出を完全に分離している点に特徴がある. |
| 任意の方向の部分空間への射影 | COP (H.-P.Kriegel, P.Kroger, E.Schubert, and A.Zimek, 2012) |
各点のk-近傍点に対してロバスト主成分分析を実行しマハラノビス距離の分布を推定.これにより,座標軸方向だけでなく,任意の方向の部分空間への射影により外れ値を検出することが可能になる. |
ELKIで外れ値検出
ELKIは,インデクス構造を用いたデータマイニングの開発・評価ツール.ルートヴィヒ・マクシミリアン大学ミュンヘンのKriegel教授を中心に開発が進められている.クラスタリング,外れ値検出のアルゴリズム,インデクスを用いたデータ構造がJavaで実装されている.
ELKIには,上記のアルゴリズムも含め,高次元データの外れ値検出のアルゴリズムが複数実装されている.Javaがインストールされた環境であれば,リリースページからjarファイルをダウンロードしてくるだけですぐに使用できる.現時点での最新版のバージョンは0.6.
以下,HiCSを例に,コマンドラインから実行し結果を確認する方法を示す.
HiCSは,以下のコマンドにより実行できる.ここでは,ELKIのウェブページで提供されているmouseデータセットを使用する*3.
$ java -jar elki.jar KDDCLIApplication -algorithm outlier.meta.HiCS -dbc.in mouse.csv -out output/meta/HiCS/mouse -lof.k 10 -hics.limit 10
algorithmオプションにHiCSを,dbc.inオプションに入力データのファイル名,outオプションに結果を格納するディレクトリを指定している.それ以降は,HiCSのパラメータである.
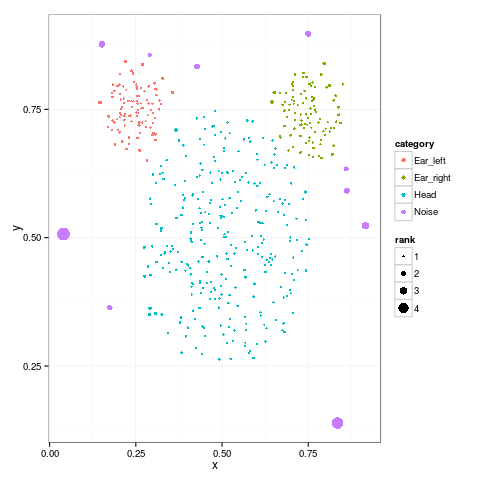
入力データのmouseデータセットは,以下のように「x座標 y座標 データが属するカテゴリ」の3つのフィールドからなる人工データである*4.
0.45660137634625386 0.43280640922410835 Head 0.6113784672224188 0.5286245988894975 Head 0.45029897412145387 0.7116061205092745 Head 0.6390150501606866 0.46074398219372076 Head 0.6289567839292338 0.32346951478531516 Head
HiCSを適用して外れ値スコアを算出するだけならば,3フィールド目の「データが属するカテゴリ」は必ずしも必要ではなく,各点の座標の値をスペース区切りで指定すれば良い.
カテゴリは"Head", "Ear_left", "Ear_right", "Noise"の4種類あり,"Noise"のカテゴリが割り当てられた点が,外れ値を意図して生成されていると思われる.
指定できるHiCSのパラメータと内容は以下の通りである.
| パラメータ | デフォルト値 | 内容 |
|---|---|---|
| hics.m | 50 | モンテカルロシミュレーションの試行回数 |
| hics.alpha | 0.1 | サンプリングパラメータ |
| hics.algo | lof.LOF | 外れ値スコアを算出するために使用するアルゴリズム |
| hics.test | KolmogorovSmirnovTest | 用いる統計的検定 |
| hics.limit | 100 | ? |
| hics.seed | null | 乱数種 |
| lof.k | なし(指定必須) | LOFを実行する際の近傍点の個数 |
上記のコマンドを実行した結果,出力先のディレクトリには以下のファイルが生成されている.
$ ls output/meta/HiCS/mouse
HiCS-outlier_order.txt head.txt pr-curve.txt settings.txt
ear_left.txt noise.txt precision-at-k.txt
ear_right.txt outlier-histogram.txt roc-curve.txt
これらのファイルは,以下の5種類に分けることができる.
- 外れ値スコアのランキング (HiCS-outlier_order.txt)
- カテゴリごとの外れ値スコアのランキング (ear_left.txt / ear_right.txt / head.txt / noise.txt)
- 外れ値検出の精度評価結果 (pr-curve.txt / precision-at-k.txt / roc-curve.txt)
- 外れ値スコアのヒストグラムのテーブル (outlier-histogram.txt)
- 外れ値検出実行時の条件 (settings.txt)
入力データにカテゴリが指定されなかった場合は,「カテゴリごとの外れ値スコアのランキング」,「外れ値検出の精度評価結果」,「外れ値スコアのヒストグラムのテーブル」の各ファイルは出力されない*5.
中核となるのは,1番目の外れ値スコアのランキングである.このランキングは,以下のように「ID=XXX X座標 Y座標 カテゴリ HiCS-outlier=外れ値スコア」というフォーマットで出力されている.外れ値スコアの上位10件は,すべてNoiseというカテゴリが付与された点であることが分かる.
$ head -n 10 output/meta/HiCS/mouse/HiCS-outlier_order.txt ID=493 0.040554927744786196 0.5072400792452862 Noise HiCS-outlier=4.990745232367167 ID=494 0.8351619113805846 0.13894038814840037 Noise HiCS-outlier=4.54623153237365 ID=499 0.9160298083819269 0.523390593285425 Noise HiCS-outlier=2.9713913373033023 ID=496 0.15150612475114178 0.8765856628207388 Noise HiCS-outlier=2.676335528483546 ID=498 0.8620825903226392 0.5918053842487218 Noise HiCS-outlier=2.4841215287717135 ID=492 0.7500676018742642 0.897027660749859 Noise HiCS-outlier=2.478742342093093 ID=500 0.42732547274373656 0.8337665738193867 Noise HiCS-outlier=2.3224593385443324 ID=495 0.17474033164278335 0.3636861115238198 Noise HiCS-outlier=2.2063046357373977 ID=497 0.8603082847506063 0.6338333996208041 Noise HiCS-outlier=2.0582792064801536 ID=491 0.290949977617754 0.8557666871174766 Noise HiCS-outlier=1.7418386083077688
このファイルをRに読み込んで,点の座標とカテゴリ,外れ値スコアを表示させてみよう.
library(ggplot2) z <- read.csv("output/meta/HiCS/mouse/HiCS-outlier_order.txt", sep=" ", header=FALSE, colClasses=c("character", "numeric", "numeric", "factor", "character")) z$V5 <- as.numeric(gsub("HiCS-outlier=", "", z$V5)) colnames(z) <- c("ID", "x", "y", "category", "rank") p <- ggplot(data=z, aes(x=x, y=y, group=category, colour=category, size=rank)) + geom_point() print(p)
カテゴリが"Head", "Ear_left", "Ear_right"のクラスタの外側に,"Noise"とカテゴリが付与された点で外れ値スコアが大きくなっていることが確認できる.
他のアルゴリズムも,algorithmオプションの引数を変更することにより実行できる.アルゴリズムと引数の対応は下表の通りである.
| アルゴリズム | algorithmオプションの引数 |
|---|---|
| ABOD | outlier.ABOD/outlier.FastABOD/outlier.LBABOD |
| Aggarwal and Yu | outlier.AggarwalYuNaive/outlier.AggarwalYuEvolutionary |
| Feature Bagging | outlier.meta.FeatureBagging |
| HiCS | outlier.meta.HiCS |
| COP | outlier.COP |
| OUTRES | outlier.subspace.OUTRES |
| OutRank | outlier.subspace.OutRankS1 |
参考文献

- 作者: Charu C. Aggarwal
- 出版社/メーカー: Springer
- 発売日: 2013/01/11
- メディア: ハードカバー
- この商品を含むブログを見る
最新の研究も含めてよくまとまっている.高次元データに限らず,データ間の空間的な近さに基づく方法(proximity-based approach),教師あり外れ値検出などについても説明されている.また,カテゴリ,テキスト,時系列,系列,空間,グラフ,ネットワークなど,幅広いデータ種別の外れ値検出にも対応.ただし,個々のアルゴリズムについては,数式や図を駆使して丁寧に説明されているわけではないので,別途,原論文等を参照する必要がある.- H.-P.Kriegel, P.Kroger, A.Zimek, Outlier Detection Techniques, The 2010 SIAM International Conference on Data Mining, 2010.
2010年のSIAMでのプレゼン資料.高次元データに限らず,距離に基づく方法(distance-based approach),密度に基づく方法(density-based approach)などについても説明がされていて,外れ値検出のイントロとして読むのに適している資料. - A.Zimek, E.Schubert, and H.-P.Kriegel, A Survey on Unsupervised Outlier Detection in High-Dimensional Numerical Data, Statistical Analysis and Data Mining,5(5):363–387,2012.
未読だが,下記のtutorialの内容を詳細に説明していると思われる.是非,機会を見つけて読みたいところ. - A.Zimek, E.Schubert, H.-P.Kriegel, Outlier Detection in High-Dimensional Data, PAKDD 2013
上記のサーベイ論文の概要を説明したtutorial.次元の呪いのイントロに始まり,高次元データの外れ値検出について一通り紹介している.この資料だけでアルゴリズムの詳細を理解するのは難しいが,最新の研究も含めて全体感を把握するのに適している.
dplyrでcolwise
dplyrを使って,plyrのcolwiseのような処理をしたい.このようなときはHadley Wickham氏がgithubで公開しているdplyrパッケージのsummarise_each関数を使用すればよい.
> library(devtools) > install_github("hadley/dplyr", ref = "colwise") > library(dplyr) > iris %.% + group_by(Species) %.% + summarise_each(funs(mean)) Source: local data frame [3 x 5] Species Sepal.Length Sepal.Width Petal.Length Petal.Width 1 setosa 5.006 3.428 1.462 0.246 2 versicolor 5.936 2.770 4.260 1.326 3 virginica 6.588 2.974 5.552 2.026
複数の集約処理も可能.
> iris %.% + group_by(Species) %.% + summarise_each(funs(min, max)) Source: local data frame [3 x 9] Species Sepal.Length_min Sepal.Width_min Petal.Length_min Petal.Width_min 1 setosa 4.3 2.3 1.0 0.1 2 versicolor 4.9 2.0 3.0 1.0 3 virginica 4.9 2.2 4.5 1.4 Variables not shown: Sepal.Length_max (dbl), Sepal.Width_max (dbl), Petal.Length_max (dbl), Petal.Width_max (dbl)
この機能はdplyr-0.2から導入予定とのこと.
参考:
How can I use dplyr to apply a function to all non-group_by columns?
冗長性が低く重要度の高いパターンの抽出(1)
パターンマイニングはデータマイニングを代表する手法の一つで,特にアソシエーションルールを適用した「ビールとおむつ」などの例が有名です.
最近は,Rなどのデータ分析ツールでもAprioriやEclat(頻出パターンマイニング), CSPADE(系列パターンマイニング)等のアルゴリズムを実行するライブラリが提供されており,パターンマイニングを実行することの障壁は比較的低くなっています.
パターンマイニングでは,一般的に膨大な数のパターンが抽出されます.この事象はアイテムの組み合わせや順列の数が膨大になることに起因しており,少量のトランザクションから大量のパターンが抽出されることも決して珍しくありません*1.このような背景の下,パターンマイニングで抽出されたパターンから重要なパターンを抽出することは,大きな技術的課題の一つだと言えるでしょう.
抽出したパターンは膨大な数に
以上で説明したことを実感するために,パターンマイニングを実行してみましょう.
FIMIレポジトリで公開されているretailデータセットに対して適用します.まず,データセットを取得します.
$ wget http://fimi.ua.ac.be/data/retail.dat # データの取得 $ wc -l retail.dat # データは88,162レコード 88162 retail.dat $ head retail.dat # データの先頭 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 38 39 47 48 38 39 48 49 50 51 52 53 54 55 56 57 58 32 41 59 60 61 62 3 39 48 63 64 65 66 67 68 32 69
FP-growthアルゴリズムにより頻出パターンを抽出します.ここでは,Christian Borgelt先生が公開されているパターンマイニングのアルゴリズムの実行ファイルの中からFP-Growthを選んで使用します.サポートを0.1%(89レコード以上)と設定して実行しましょう.
$ # 最小アイテム数2, サポート0.001(0.1%), アイテム間のセパレータ","(-k,), アイテムセットの出現指標はサポート $ ./fpgrowth -m2 -s0.1 -k, -v" %s" retail.dat fpgrowth_retail.dat $ wc -l fpgrowth_retail.dat # サポート0.1%以上のアイテムセットは5,472個 5472 fpgrowth_retail.dat $ head fpgrowth_retail.dat # FP-growthを用いているためアイテムセットはサポート順に並ぶとは限らない 48,39 0.330551 38,39 0.117341 38,48,39 0.0692135 38,48 0.0901068 32,39 0.095903 32,48,39 0.0612736 32,48 0.0911277 32,38,39 0.0208707 32,38,48,39 0.0140196 32,38,48 0.0186702 $ sort -k2 -r fpgrowth_retail.dat | head # サポートの降順にアイテムセットをソート 48,39 0.330551 41,39 0.129466 38,39 0.117341 41,48 0.102289 32,39 0.095903 32,48 0.0911277 38,48 0.0901068 41,48,39 0.0835507 38,48,39 0.0692135 32,48,39 0.0612736
大量のパターンが抽出されてしまいました・・・
冗長性が低く重要度が高いパターンを抽出する
さて,このように膨大な数のパターンが抽出されるパターンマイニングの結果から,なるべく少なく情報量が多いパターンを列挙するためにはどうすれば良いでしょうか.このような問題意識から,いくつかの研究が行われています.この中から,今回は,冗長性が低く重要度が高いパターンを抽出することを目指して,以下のKDD'06の論文を取り上げます.
D.Xin, H.Cheng, X.Yan, J.Han, Extracting redundancy-aware top-k patterns, KDD'06.
この論文で紹介されている手法は,最初から一般論だと少し分かりづらい面もあるかもしれないので,次の小さなデータセットを例にとって説明しましょう.
test.dat
a b c a d e b c d a b c d b c a b d d e a b c d c d e a b c
このデータセットに対して,上記と同様の手続きによってパターンマイニングを実行すると,以下の結果を得ます.行頭の連番は以後の説明のために付与したものです.
1. c,b 0.6 2. d,c 0.4 3. d,b,c 0.3 4. d,b 0.4 5. a,b 0.5 6. a,d,b 0.3 7. a,d 0.4 8. a,c,b 0.4 9. a,c 0.4 10. e,d 0.3
この中から,5個だけパターンを選びましょう.単純にサポートが大きいパターンの順に選ぶと,以下の7つが候補となるでしょう.
- 1. c,b 0.6
- 5. a,b 0.5
- 2. d,c 0.4
- 4. d,b 0.4
- 7. a,d 0.4
- 8. a,c,b 0.4
- 9. a,c 0.4
末尾にサポート0.4のパターンが5つ並んでおり,この方法では選び出す5個のパターンを決定できません.
少し見方を変えて,各パターンが元々のトランザクションデータのどのレコードに出現していたかについて調べてみましょう.
1. c,b 0.6 1, 3, 4, 5, 8, 10 2. d,c 0.4 3, 4, 8, 9 3. d,b,c 0.3 3, 4, 8 4. d,b 0.4 3, 4, 6, 8 5. a,b 0.5 1, 4, 6, 8, 10 6. a,d,b 0.3 4, 6, 8 7. a,d 0.4 2, 4, 6, 8 8. a,c,b 0.4 1, 4, 8, 10 9. a,c 0.4 1, 4, 8, 10 10. e,d 0.3 2, 7, 9
3フィールド目が出現したレコード番号のリストです.これを見ると,例えば
- 「8. a,c,b」と「9. a,c」はどちらも番号1, 4, 8, 10のレコードに出現している.
- サポートが最も高い「1. c,b」と2番目の「5. a,b」は,番号1, 4, 8, 10のレコードで共通に出現している.
といったことが分かります.
1番目の考察から,「9. a,c」が出現するときは,さらにbも追加された「8. a,c,b」が必ず出現していることが分かります.また,2番目の考察から,サポートがトップ2のパターンも,異なるトランザクションで独立に現れるのではなく,トランザクションの共通部分があることが分かります.
このように,2つのパターンの間でのトランザクションの重なりがあることが,パターンが独立ではなく,お互いに何かしらの関係を持つことになります.この関係は,今回取り上げた論文では「冗長性」(redundancy)という概念により定量化されます.
冗長性はどのように測れば良いでしょうか.論文では,
冗長性 = (2つのパターン間の距離) × (2つのパターンの重要性の最小値)
- 「1. c.b」と「5. a,b」は,レコード1, 3, 4, 5, 6, 8, 10でどちらか一方が現れ,レコード1, 4, 8, 10で共通に現れている.⇒ 冗長性は,距離
最小サポート
.
- 「8. a,c,b」と「9. a,c」は,レコード1, 4, 8, 10で共通に現れている.⇒ 冗長性は,距離
最小サポート
.
となります.
さて,サポート等のパターン間の重要性が与えられ,冗長性が定義できたので,選び出したパターンの集合に対して次のように評価指標を定義できるでしょう.
パターンの集合の評価指標 = 各パターンの重要性の和 - パターン間の冗長性の和
Redundancy-aware Top-k Patternの定式化
以上で説明した方法を定式化すると,次のようになります.
パターンの集合の評価
個のパターンの集合
の有益さを評価してみましょう.前提として,各パターンの重要度
が求められているとします.重要度の指標として,サポート,コンフィデンス,リフトなどが用いられます.
各パターンが独立の場合は,評価関数は各パターンの重要度の和,すなわち,
で定義できるでしょう.
しかし,一般的にはパターン間には冗長性があるため,評価関数は
MASとMMS
上記のパターン集合に対する評価の定式化においては,冗長性をどのように定義するかがポイントになるます.本論文では,MAS(Maximal Average Significance, 極大平均重要度)とMMS(Maximal Marginal Significance)の2つが提案されています.以下ではパターンの個数
は外から指定するものとします.
- MAS(Maximal Average Significance)
以下で定義する指標が最大となる
個のパターンの集合
を抽出する.
ここで,はパターン
と
の間の冗長性であり,パターン間の距離
を用いて次式で定義される.
- MMS(Maximal Marginal Significance)
以下で定義する指標が最大となる
個のパターンの集合
を抽出する.
MASもMMSも最適解を求めることはNP-hardであるため,逐次的に近似解を求めていきます.個目までのパターンが抽出されたとき,パターン
に対して,それまでに抽出されたパターンとの冗長性を求めることにより
を採用することによるゲイン
を評価します.すなわち,ゲイン
を次式
上式を用いて個のパターンを抽出すると計算量は
となります.しかし,
回目までに抽出されなかったパターンに対しては,
回目に算出したゲインを用いてインクリメンタルに
回目のゲインを計算することにより,計算量を
に削減できます.すなわち,パターン
に対して以下のように
回目のゲインの結果
を用いて
回目のゲイン
を算出します.
適用例
上記のretailデータセットの頻出パターンに対して,Top-Kのパターンを抽出してみましょう.
- 入力ファイル: fpgrowth_retail.dat
- トランザクションデータファイル: retail.dat
- 抽出するパターン数: 10
- 出力ファイル: redtopk_retail.dat
$ ./redtopk -i fpgrowth_retail.dat -t retail.dat -k 10 -o redtopk_retail.dat
以下の結果が得られます.
$ sort -k2 -r redtopk_retail.dat
{39,48} 0.330551
{39,41} 0.129466
{38,39} 0.117341
{41,48} 0.102289
{32,39} 0.095903
{32,48} 0.0911277
{38,48} 0.0901068
{39,41,48} 0.0835507
{38,39,48} 0.0692135
{32,39,48} 0.0612736
論文の後半では,MMSに対してグラフを用いて計算を効率化するアルゴリズムが提案されています.それについてはまた別の記事で書きたいと思います.
参考文献

- 作者: Jiawei Han,Jian Pei,Micheline Kamber
- 出版社/メーカー: Morgan Kaufmann
- 発売日: 2011/06/09
- メディア: Kindle版
- この商品を含むブログを見る
*1:もちろん,supportやconfidenceの値の設定に大きく依存します
頻出アイテムセット間のJaccard係数の計算
Jaccard係数(Jaccard index, Jaccard similarity coefficent)は,2つの集合間の類似性を表す指標.
パターンマイニングでは,2つの頻出パターンの共起を表す指標として用いられ,両方のパターンが現れるトランザクション数に対して,少なくとも一方のパターンが現れるトランザクション数の割合として定義される.すなわち,パターンに対して,Jaccard係数
は,
以下のテストデータを用意して,頻出パターン,特にここでは頻出アイテムセットに対するJaccard係数を算出してみよう.
test.dat
a b c a d e b c d a b c d b c a b d d e a b c d c d e a b c
頻出アイテムセットの抽出
上記のデータに対してアソシエーションルールを実行して頻出アイテムセットを抽出する.ここでは,Christian Borgelt先生が公開されているパターンマイニングのアルゴリズムの実行ファイルからFP-Growthを選択する.
$ # 実行ファイルの取得 $ wget http://www.borgelt.net//bin64/fpgrowth $ # 最小アイテム数2, サポート0.3, アイテム間のセパレータ","(-k,), アイテムセットの出現指標は出現数 $ ./fpgrowth -m2 -s30 -k, -v" %n" test.dat test.out
以下の10個の頻出パターンが"test.out"に出力される.この出力されたデータは,頻出アイテムセットとその出現回数に関する情報を保持していて,例えば
- アイテムセット {c,b} が6回出現
- アイテムセット {d,c} が4回出現
といったことが分かる.
test.out
c,b 6 d,c 4 d,b,c 3 d,b 4 a,b 5 a,d,b 3 a,d 4 a,c,b 4 a,c 4 e,d 3
Jaccard係数の算出
続いて,Jaccard係数を算出する.main関数を見れば分かるように,処理は大きく4つのステップ
- トランザクションデータの読み込み
- 頻出アイテムセットの読み込み
- 頻出アイテムセットとトランザクションデータのマッチング → 各アイテムセットが含まれるトランザクション番号の抽出
- アイテムセット間のJaccard係数の算出
からなる.
#include <iostream> #include <sstream> #include <fstream> #include <vector> #include <string> #include <map> #include <algorithm> #include <iterator> #include <boost/tokenizer.hpp> #include <boost/algorithm/string/join.hpp> using namespace std; // コンテナの定義 typedef vector<string> Tran; // トランザクション typedef vector<Tran> TranSet; // トランザクションの集合 typedef vector<string> Pattern; // パターン typedef vector<Pattern> PatternSet; // パターンの集合 typedef size_t tranid; // トランザクションID typedef map<Pattern, vector<tranid>> PatTranSet; // 頻出パターンとトランザクションIDの対応 // トークナイザの設定 typedef boost::char_separator<char> char_separator; typedef boost::tokenizer<char_separator> tokenizer; /** * @brief ファイルからトランザクションデータを読み込む * @param fn トランザクションデータのファイル名 * @param trset トランザクションデータを格納するコンテナ */ void readTran(string fn, TranSet &trset) { string line; // 入力データのレコード Tran tr; // トランザクション string item; // アイテム ifstream ifs(fn.c_str()); // 各トランザクションの読み込み while (ifs && getline(ifs, line)) { tr.clear(); istringstream istrs((char *) line.c_str()); // 各アイテムの読み込み while (istrs >> item) { // 同一のトランザクションで同一のアイテムはユニークにする vector<string>::iterator itr_tr; if (find(tr.begin(), tr.end(), item) == tr.end()) { tr.push_back(item); } } sort(tr.begin(), tr.end()); trset.push_back(tr); } } /** * @brief ファイルから頻出アイテムセットを読み込む * @param fn 頻出アイテムセットのファイル名 * @param patset 頻出アイテムセットを格納するコンテナ * @param sep_pat 頻出アイテムセットとその他の情報のセパレータ(頻出アイテムセットの位置が第1フィールドであることが前提) * @param sep_item 頻出アイテムセットのアイテム間のセパレータ */ void readPattern(string fn, PatternSet &patset, string sep_pat=" ", string sep_item=",") { string line; // 入力データのレコード Pattern pat; // 頻出アイテムセット string item; // アイテム ifstream ifs(fn.c_str()); char_separator separator(sep_pat.c_str(), "", boost::keep_empty_tokens); // 各アイテムセットの読み込み while (ifs && getline(ifs, line)) { pat.clear(); // 各アイテムへの分割と格納 tokenizer tokens(line, separator); string str = *tokens.begin(); istringstream istrs((char *) str.c_str()); while (std::getline(istrs, item, ',')) { pat.push_back(item); } sort(pat.begin(), pat.end()); patset.push_back(pat); } } /** * @brief 一方のベクタが他方のベクタの部分集合になっているかについて調べる(a is a subset of b) * @param a 他方のベクタからの被包含を調べる対象のベクタ * @param b 他方のベクタの包含を調べる対象のベクタ */ template <typename T> bool isSubset(vector<T> a, vector<T> b) { return includes(b.begin(), b.end(), a.begin(), a.end()); } /** * @brief 頻出アイテムセットとトランザクションデータをマッチングして,各アイテムセットが含まれるトランザクション番号を抽出する * @param ps 頻出アイテムセットを格納するコンテナ * @param ts トランザクションを格納するコンテナ * @param pts 頻出アイテムセットが含まれるトランザクション番号を格納するコンテナ */ void patmatch(PatternSet& ps, TranSet& ts, PatTranSet & pts) { // 各アイテムセットに対するトランザクションデータとのマッチング PatternSet::iterator ps_itr = ps.begin(); while (ps_itr != ps.end()) { tranid tn = 1; // トランザクション番号(1からの連番) vector<tranid> tn_set; // トランザクション番号を格納するコンテナ // 各トランザクションとのマッチング TranSet::iterator tr_itr = ts.begin(); while (tr_itr != ts.end()) { bool matched = isSubset(*tr_itr, *ps_itr); // マッチした場合はトランザクション番号を格納する if (matched) { tn_set.push_back(tn); } tr_itr++; ln++; } pts.insert(map<Pattern, vector<tranid>>::value_type(*ps_itr, ln_set)); ps_itr++; } } /** * @brief 2つの頻出アイテムセット間のjaccard係数を算出する * @param p1 頻出アイテムセット * @param p2 頻出アイテムセット */ double jaccard(const Pattern &p1, const Pattern &p2) { // アイテムセットの共通集合を求める Pattern intersec; set_intersection(p1.begin(), p1.end(), p2.begin(), p2.end(), back_inserter(intersec)); // Jaccard係数を算出する int size_intersec = intersec.size(); int size_union = p1.size() + p2.size() - size_intersec; return (double)size_intersec/size_union; } /** * @brief 与えられた全ての頻出アイテムセット間のJaccard係数を算出する * @param pts 各頻出アイテムセットのトランザクション番号を保持したコンテナ */ void jaccard_all(const PatTranSet &pts) { // 各頻出アイテムセットに対して他のアイテムセットとのJaccard係数を算出する PatTranSet::const_iterator itr1 = pts.begin(); while (itr1 != pts.end()) { Pattern pat1 = itr1->first; string p1 = "{" + boost::algorithm::join(pat1, ",") + "}"; // Jaccard係数を算出する対象のアイテムセットを抽出する PatTranSet::const_iterator itr2 = ++pts.begin(); while (itr1 != itr2 && itr2 != pts.end()) { Pattern pat2 = itr2->first; string p2 = "{" + boost::algorithm::join(pat2, ",") + "}"; // Jaccard係数を算出する cout << " Jaccard(" << p1 << "," << p2 << ") = " << jaccard(pat1, pat2) << endl; itr2++; } itr1++; } } int main() { // 1. トランザクションデータの読み込み string fn_tran="test.dat"; TranSet trset; cout << "Reading transaction: " << fn_tran << endl; readTran(fn_tran, trset); // 2. 頻出アイテムセットの読み込み string fn_pat="test.out"; string sep_pat=" "; string sep_item=","; PatternSet patset; cout << "Reading frequnet itemset: " << fn_pat << endl; readPattern(fn_pat, patset, sep_pat, sep_item); // 3. 頻出アイテムセットとトランザクションデータのマッチング PatTranSet pts; cout << "Pattern matching" << endl; patmatch(patset, trset, pts); // 4. アイテムセット間のJaccard係数の算出 cout << "Calculating Jaccard coefficient" << endl; jaccard_all(pts); return 0; }
以上のプログラムをコンパイル,実行してみよう.Boostライブラリは/usr/includeに入っているものとする.
$ g++ -Wall -std=c++11 -I /usr/include -o patsim patsim.cpp
$ ./patsim
Reading transaction: test.dat
Reading frequnet itemset: test.out
Pattern matching
Calculating Jaccard coefficient
Jaccard({a,b},{a,b,c}) = 0.666667
Jaccard({a,b},{a,b,d}) = 0.666667
Jaccard({a,b},{a,c}) = 0.333333
Jaccard({a,b},{a,d}) = 0.333333
Jaccard({a,b},{b,c}) = 0.333333
Jaccard({a,b},{b,c,d}) = 0.25
Jaccard({a,b},{b,d}) = 0.333333
Jaccard({a,b},{c,d}) = 0
Jaccard({a,b},{d,e}) = 0
Jaccard({a,b,d},{a,b,c}) = 0.5
Jaccard({a,c},{a,b,c}) = 0.666667
Jaccard({a,c},{a,b,d}) = 0.25
Jaccard({a,d},{a,b,c}) = 0.25
Jaccard({a,d},{a,b,d}) = 0.666667
Jaccard({a,d},{a,c}) = 0.333333
Jaccard({b,c},{a,b,c}) = 0.666667
Jaccard({b,c},{a,b,d}) = 0.25
Jaccard({b,c},{a,c}) = 0.333333
Jaccard({b,c},{a,d}) = 0
Jaccard({b,c,d},{a,b,c}) = 0.5
Jaccard({b,c,d},{a,b,d}) = 0.5
Jaccard({b,c,d},{a,c}) = 0.25
Jaccard({b,c,d},{a,d}) = 0.25
Jaccard({b,c,d},{b,c}) = 0.666667
Jaccard({b,d},{a,b,c}) = 0.25
Jaccard({b,d},{a,b,d}) = 0.666667
Jaccard({b,d},{a,c}) = 0
Jaccard({b,d},{a,d}) = 0.333333
Jaccard({b,d},{b,c}) = 0.333333
Jaccard({b,d},{b,c,d}) = 0.666667
Jaccard({c,d},{a,b,c}) = 0.25
Jaccard({c,d},{a,b,d}) = 0.25
Jaccard({c,d},{a,c}) = 0.333333
Jaccard({c,d},{a,d}) = 0.333333
Jaccard({c,d},{b,c}) = 0.333333
Jaccard({c,d},{b,c,d}) = 0.666667
Jaccard({c,d},{b,d}) = 0.333333
Jaccard({d,e},{a,b,c}) = 0
Jaccard({d,e},{a,b,d}) = 0.25
Jaccard({d,e},{a,c}) = 0
Jaccard({d,e},{a,d}) = 0.333333
Jaccard({d,e},{b,c}) = 0
Jaccard({d,e},{b,c,d}) = 0.25
Jaccard({d,e},{b,d}) = 0.333333
Jaccard({d,e},{c,d}) = 0.333333Jaccard係数を算出できていることが分かる.
ただし,上記のコードにはいろいろと改善の余地がある.例えば,
- パターンマイニングを実行して頻出アイテムセットを抽出した後,改めてトランザクションデータとのマッチングを行っているため計算効率が悪い*1.
- 仮にトランザクションデータのマッチングを行う方針でも,全件マッチングとなっているため計算効率が悪い.
などである.改善方法については別途記事を書きたいところ.
knitr+LaTeXでPDFを作成するmakefile
以前,TokyoRで「RでReproducible Research」というタイトルの発表を行いました.Reproducible Researchとは再現性のある研究のことで,そのためには処理に再現性が担保されている必要があります.
RでReproducible Researchを実現する上で,動的なレポート生成を行うknitrパッケージが有力なツールになると考えられます.knitrを用いることにより,プログラムとドキュメントを同時に記述することが可能になります.このようにして,使用したデータや処理を明確にした上で,多様な形式のレポートを作成することができるようになります.knitrの詳細については,作者による本家のホームページ,浅井さんのページが詳しいのでご参照ください.
LaTeXでPDFファイルをレポートとして作成する場合は,次のようなmakefileを用意することにより,コマンド一発でRmdファイル → texファイル → dviファイル → pdfファイルまで作成できるようになります.
.SUFFIXES: .Rnw .tex .dvi .pdf
RSCRIPT=/usr/bin/Rscript
EXEDIR=/usr/local/bin
LATEX=$(EXEDIR)/platex
BIBTEX=/usr/bin/bibtex
MKIDX=$(EXEDIR)/makeindex
DVIPDF=$(EXEDIR)/dvipdfmx
.Rnw.tex:
$(RSCRIPT) -e "library(knitr); knit('$<')"
.tex.dvi:
$(LATEX) $<
$(BIBTEX) $*
$(MKIDX) $<
$(LATEX) $<
$(LATEX) $<
.dvi.pdf:
$(DVIPDF) $<
target=test
all: ${target}.pdf
clean:
rm -f *~
rm -f *.{aux,bbl,blg} *.log ${target}.{lof,lot,toc,tex,dvi,pdf,out}
Dynamic Documents with R and knitr (Chapman & Hall/CRC The R Series)
- 作者: Yihui Xie
- 出版社/メーカー: Chapman and Hall/CRC
- 発売日: 2013/09/10
- メディア: ペーパーバック
- この商品を含むブログを見る
Rcppによるbigmemoryの拡張
RのbigmemoryパッケージはC++で実装されているため,ユーザが新たな機能を開発して追加することが可能です.Rcpp Galleryの"Using bigmemory with Rcpp"(各列の合計値を算出する例)をそのまま実行してみます.
BigColSums.cpp
#include <Rcpp.h> // [[Rcpp::depends(bigmemory)]] #include <bigmemory/MatrixAccessor.hpp> #include <numeric> // [[Rcpp::export]] Rcpp::NumericVector BigColSums(Rcpp::XPtr<BigMatrix> pBigMat) { // big.matrix型の行列へのアクセッサ MatrixAccessor<double> ma(*pBigMat); // 各列の合計値を格納する数値型ベクトル Rcpp::NumericVector colSums(pBigMat->ncol()); // 各列の合計値を算出する for (size_t i=0; i < pBigMat->ncol(); ++i) colSums[i] = std::accumulate(ma[i], ma[i]+pBigMat->nrow(), 0.0); return colSums; }
Rを起動して以下を実行します.
> suppressMessages(library(bigmemory)) > library(Rcpp) > # C++ソースのコンパイル > sourceCpp("BigColSums.cpp") > # bigmemoryのバッキングファイル,ディスクリプタファイル > bkFile <- "bigmat.bk" > descFile <- "bigmatk.desc" > # big.matrix型行列の作成(10,000×3) > nrows <- 10000 > bigmat <- filebacked.big.matrix(nrow=nrows, ncol=3, type="double", + backingfile=bkFile, backingpath=".", + descriptorfile=descFile, + dimnames=c(NULL,NULL)) > > set.seed(123) > # 各列に正規乱数を格納 > for (i in 1:3) bigmat[,i] = rnorm(nrows)*i > # 各列の合計値を算出 > res <- BigColSums(bigmat@address) > res [1] -23.71702 -182.12906 -212.98229
各列の合計値が算出されたことを確認できます.